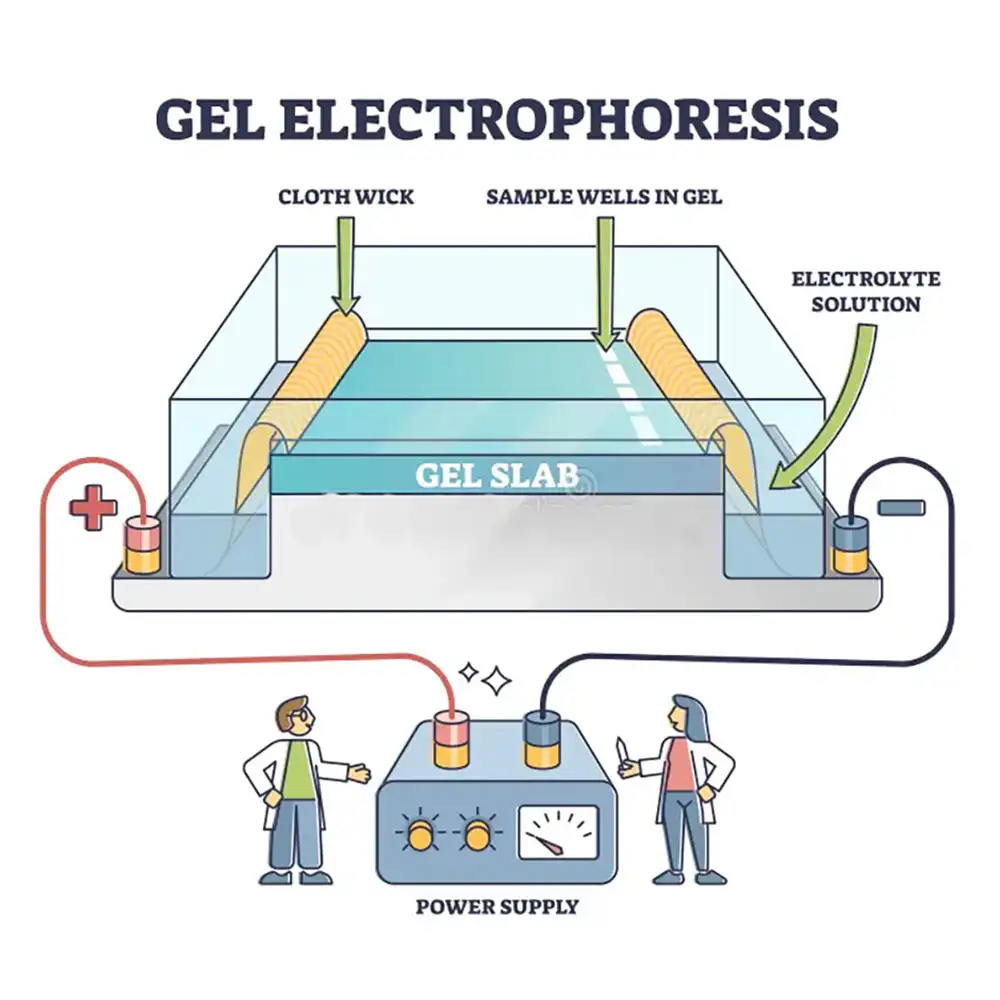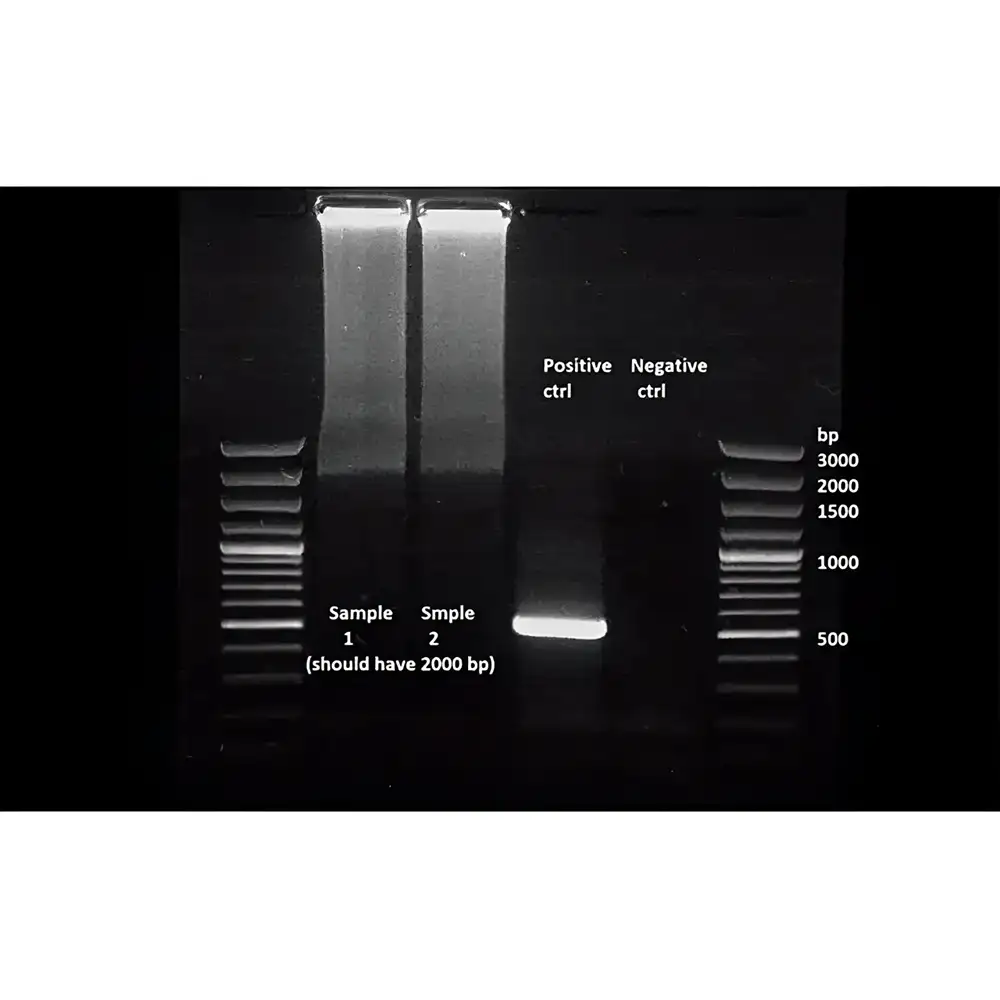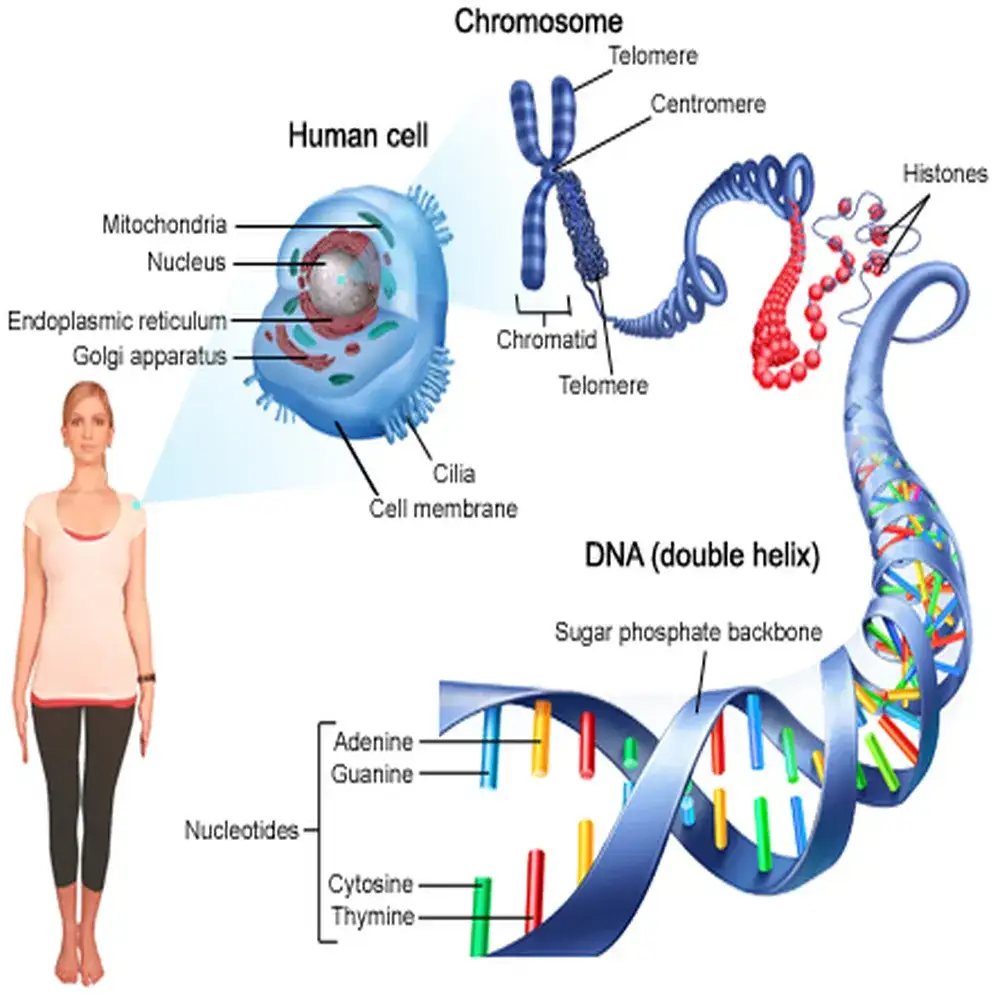
ترانسکریپتومیکس چیست
فصل اول: مقدمهای بر ترنسکریپتومیکس
ترنسکریپتومیکس یکی از شاخههای زیستشناسی مولکولی است که بر مطالعهی جامع تمام مولکولهای RNA تولیدشده در یک سلول یا بافت، در شرایط خاص، تمرکز دارد. این علم با تحلیل مجموعهی کامل RNAها که به طور کلی با عنوان «ترنسکریپتوم» شناخته میشود، درک عمیقی از بیان ژنها ارائه میدهد.
برخلاف ژنوم که ساختاری نسبتاً ثابت دارد، ترنسکریپتوم پویا است و بسته به محرکهای داخلی و خارجی تغییر میکند. این ویژگی پویایی، ترنسکریپتومیکس را به ابزاری قدرتمند برای ثبت آنی عملکرد سلولی تبدیل کرده است.
ترنسکریپتوم چیست؟
ترنسکریپتوم شامل تمام انواع RNA است که از ژنوم رونویسی میشوند، چه آنهایی که کد پروتئین دارند و چه آنهایی که فاقد چنین نقشی هستند. این RNAها شامل موارد زیر میشوند:
-
RNA پیامرسان (mRNA): این مولکولها دستورالعملهای ژنتیکی را از DNA به ریبوزومها منتقل میکنند، جایی که ترجمه به پروتئین انجام میشود. مقدار mRNA نشاندهندهی ژنهایی است که در یک زمان مشخص در حال بیان فعال هستند.
-
RNA ریبوزومی (rRNA): بخش ساختاری و عملکردی ریبوزوم است و برای فرآیند ترجمهی mRNA به پروتئین ضروری میباشد.
-
RNA ناقل (tRNA): آمینواسیدهای مشخص را طی سنتز پروتئین به ریبوزوم منتقل میکند و هر کدون موجود در mRNA را با آمینواسید مناسب تطبیق میدهد.
-
RNAهای غیرکدکننده (ncRNAs): گروهی متنوع از RNAها هستند که پروتئینی تولید نمیکنند اما نقشهای تنظیمی بسیار مهمی دارند. این گروه شامل microRNA (miRNA)، small interfering RNA (siRNA)، long non-coding RNA (lncRNA) و سایر انواع RNAهای دخیل در خاموشسازی ژنها، بازآرایی کروماتین، پیرایش RNA و سایر عملکردهای تنظیمی میباشد.
چرا ترنسکریپتومیکس اهمیت دارد؟
ترنسکریپتوم بازتاب مستقیمی از فعالیت ژنها ارائه میدهد. در حالی که DNA نقشهی پایهی عملکرد زیستی را فراهم میکند، RNA نشان میدهد که این نقشه در زمان واقعی چگونه به کار گرفته میشود. ترنسکریپتومیکس این امکان را فراهم میسازد تا دانشمندان بتوانند:
-
بررسی کنند که کدام ژنها در شرایط مختلف (مانند بافت سالم در برابر بافت بیمار) فعال یا غیرفعال هستند.
-
مطالعه کنند که سلولها چگونه به سیگنالهای بیرونی، استرس، داروها یا تغییرات محیطی پاسخ میدهند.
-
RNAهای جدیدی را کشف کنند که پیشتر قابلشناسایی نبودهاند.
-
بیان ژن ویژهی هر نوع سلول را بررسی کرده و به درک بهتری از رشد، تمایز و بیماری دست یابند.
ماهیت پویای ترنسکریپتوم
یکی از ویژگیهای کلیدی ترنسکریپتوم، تغییرات زمانی و مکانی آن است. بیان ژنها نهتنها در انواع مختلف سلول، بلکه در یک نوع سلول واحد نیز در زمانها یا شرایط مختلف متفاوت است. به همین دلیل، ترنسکریپتومیکس برای اهداف زیر بسیار ضروری است:
-
مطالعات زمانمند، که در آن بیان ژنها در نقاط زمانی مختلف اندازهگیری میشود.
-
تحلیلهای مقایسهای، مانند مقایسهی سلولهای طبیعی با سلولهای سرطانی.
-
مطالعات زیستشناسی رشد، جایی که الگوهای بیان ژن در طول عمر یک موجود زنده تغییر میکنند.
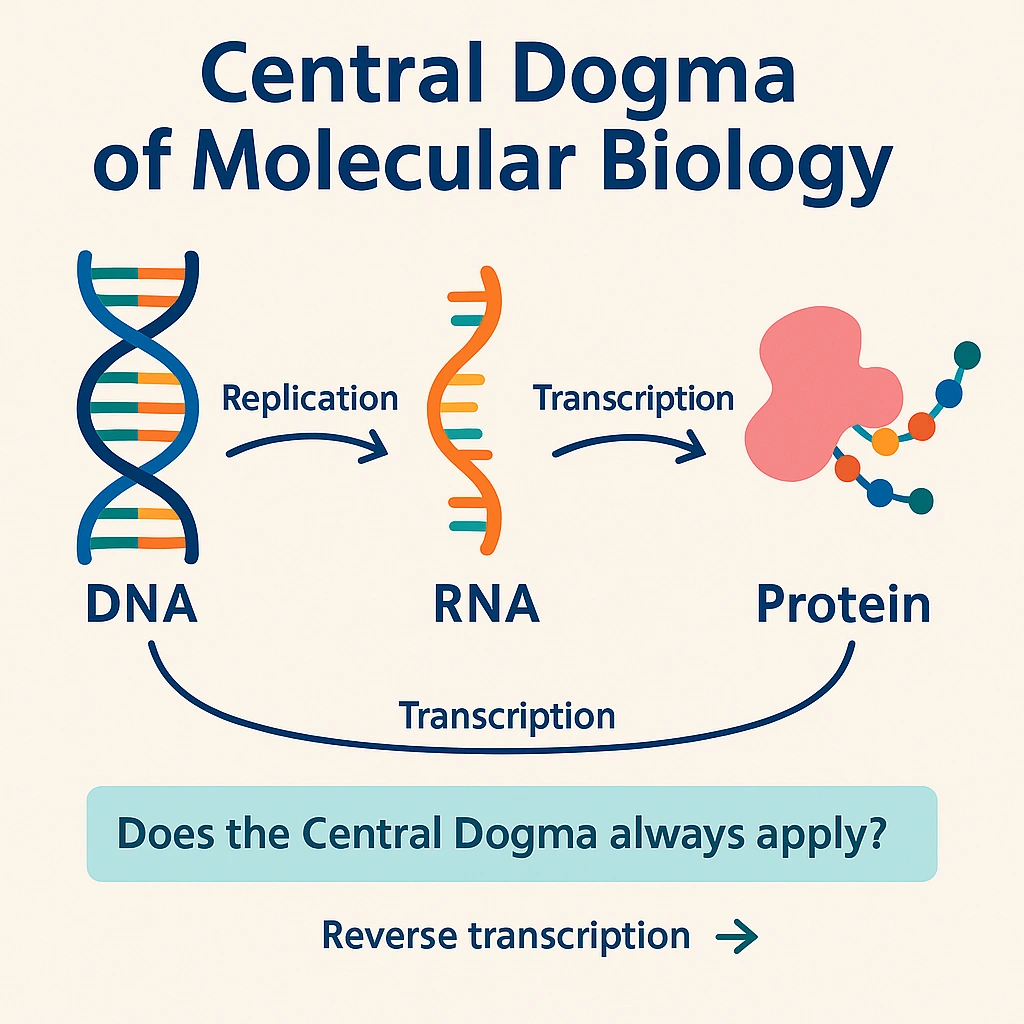
از ژن تا ترنسکریپت: آموزهی اصلی زیستشناسی مولکولی
درک ترنسکریپتومیکس نیازمند آشنایی با دکترین مرکزی زیستشناسی مولکولی است که میگوید:
DNA → RNA → پروتئین
ژنوم به ما نشان میدهد که چه ژنهایی وجود دارند، در حالی که ترنسکریپتومیکس نشان میدهد کدامیک از این ژنها فعالانه RNA تولید میکنند، که مرحلهی واسطهای پیش از سنتز پروتئین است.
بنابراین، ترنسکریپتومیکس پلی است میان ژنوم (پتانسیل) و پروتئوم (عملکرد واقعی).
دامنهی تحقیق در ترنسکریپتومیکس
تحقیقات در ترنسکریپتومیکس در سطوح مختلفی انجام میشود:
-
تحلیل RNA به صورت حجمی (Bulk RNA Analysis): RNA از جمعیتی از سلولها یا یک بافت استخراج شده و بهصورت میانگین بررسی میشود.
-
ترنسکریپتومیکس تکسلولی (Single-cell Transcriptomics): بیان ژن در سطح سلول منفرد بررسی میشود و امکان شناسایی ناهمگونی درون جمعیت سلولی و یافتن زیرجمعیتهای نادر را فراهم میکند.
-
ترنسکریپتومیکس عملکردی (Functional Transcriptomics): در این روش، دادههای بیان ژن با ویژگیهای فنوتیپی یا فرآیندهای زیستی خاص مرتبط میشوند.
فناوریها و ابزارها
پیشرفتهای فناورانه موجب شدهاند که ترنسکریپتومیکس بهطور روزافزونی قدرتمند و در دسترس شود. روشهای رایج شامل موارد زیر هستند:
-
میکروآری (Microarrays): از اتصال (Hybridization) برای شناسایی توالیهای RNA شناختهشده استفاده میکند.
-
RNA-Seq: روشی بدون تعصب، کمّی، و با توان بالا برای تحلیل کل ترنسکریپتوم است.
-
qRT-PCR: بهعنوان روشی دقیق برای اعتبارسنجی تغییرات بیان ژن بهکار میرود.
-
RNA-Seq تکسلولی: امکان تحلیل بیان ژن در سطح سلول منفرد را فراهم میسازد.
هرکدام از این تکنیکها مزایای خاص خود را دارند و بر اساس نوع نمونه، سؤال تحقیقاتی، و میزان دقت موردنیاز انتخاب میشوند.
فصل دوم: توسعه تاریخی ترنسکریپتومیکس
علم ترنسکریپتومیکس طی چند دههی گذشته، دگرگونی چشمگیری را پشت سر گذاشته است. از کشف ابتدایی مولکولهای RNA تا توسعهی فناوریهای پیشرفتهی توالییابی با توان بالا (High-throughput)، تاریخچهی ترنسکریپتومیکس بازتابی از پیشرفتهای گستردهتر در زمینههای زیستشناسی مولکولی، بیوشیمی و ژنومیکس است. درک این روند تحول، برای شناخت تواناییها و محدودیتهای تکنیکهای ترنسکریپتومیکی امروزی اهمیت زیادی دارد.
آغاز داستان: دهه ۱۹۶۰
ماجرای ترنسکریپتومیکس در دهه ۱۹۶۰ آغاز شد، زمانی که پژوهشگران مولکول RNA پیامرسان (mRNA) را به عنوان واسطهای میان DNA و سنتز پروتئین کشف کردند. این کشف، تأییدی کلیدی بر آموزهی اصلی زیستشناسی مولکولی بود که بیان میکند اطلاعات ژنتیکی از DNA به RNA و سپس به پروتئین منتقل میشود.
مطالعات اولیه بر روی mRNA عمدتاً نقش آن در انتقال دستورالعملهای ژنتیکی از هسته به سیتوپلاسم را بررسی میکردند. این تحقیقات، پایهگذار مفهومی به نام تنظیم بیان ژن شدند.
دهههای ۱۹۷۰ و ۱۹۸۰: آغاز روشهای تحلیلی
در این دوره، چندین روش برای تحلیل مولکولهای RNA توسعه یافت، اما اغلب این روشها پوشش محدودی داشتند و توان عملیاتی آنها پایین بود.
-
یکی از اولین و تأثیرگذارترین روشها، تکنیک نورتِرن بلات (Northern blotting) بود. این روش برای شناسایی توالیهای خاص RNA در میان ترکیبی پیچیده از RNAها استفاده میشد.
گرچه نورتِرن بلات اطلاعات ارزشمندی دربارهی اندازه و میزان RNA ارائه میداد، اما زمانبر، دشوار و محدود به بررسی تعداد کمی از ژنها بود. -
روشهای دیگری مانند RT-PCR (واکنش زنجیرهای پلیمراز با رونویسی معکوس) نیز توسعه یافتند. این تکنیکها حساسیت بالاتری داشتند اما همچنان در مقیاسپذیری و گسترهی کاربرد محدود بودند.
دهه ۱۹۹۰: انقلابی به نام میکروآری (Microarray)
تحولی اساسی در دههی ۱۹۹۰ رخ داد؛ با معرفی فناوری DNA Microarray. این فناوری امکان اندازهگیری همزمان سطح بیان هزاران ژن را فراهم کرد.
میکروآریها بر اساس اتصال RNA یا cDNA نشاندار شده به پروبهای DNA مکمل تثبیتشده بر روی یک سطح جامد کار میکنند. شدت فلورسانس حاصل از این اتصال، با میزان هر ترنسکریپت (RNA خاص) رابطه مستقیم دارد.
این فناوری، تحقیقات ترنسکریپتومیکی را متحول کرد و امکان مقایسههای گستردهی بیان ژنها در نمونههای زیستی مختلف، شرایط گوناگون، و بیماریها را فراهم نمود.
میکروآریها بهسرعت به ابزار استاندارد در ژنومیکس کاربردی و زیستشناسی سامانهای (Systems Biology) تبدیل شدند.
محدودیتهای میکروآری و نیاز به تحول جدید
با وجود نوآوریهای بزرگ، میکروآریها محدودیتهای ذاتی داشتند:
-
نیاز به دانستن توالی ژنهای هدف از قبل، که استفاده را محدود به ژنهای شناختهشده میکرد.
-
مشکلاتی مانند اتصال غیرتخصصی (cross-hybridization) و دامنهی دینامیکی محدود که دقت دادهها را تحت تأثیر قرار میداد.
این چالشها زمینهساز گام بعدی در تحول ترنسکریپتومیکس شدند: توالییابی RNA یا RNA-Seq.
دهه ۲۰۰۰: ظهور RNA-Seq و انقلاب NGS
با توسعهی فناوریهای توالییابی نسل جدید (NGS) در اوایل دهه ۲۰۰۰، عصر جدیدی در ترنسکریپتومیکس آغاز شد.
RNA-Seq بهعنوان جایگزینی قدرتمند برای میکروآریها معرفی شد و تواناییهایی بسیار فراتر ارائه داد:
-
غیرجانبدارانه (Unbiased): بدون نیاز به پروبهای از پیش طراحیشده.
-
با دقت بالا، کمّی و در مقیاس وسیع: امکان شناسایی ترنسکریپتهای شناختهشده و ناشناخته.
-
قابلیت تشخیص وقایع پیچیدهای مانند:
-
Alternative splicing (اتصال جایگزین اگزونها)
-
Fusion genes (ادغام ژنها)
-
RNAهای غیرکدکننده
-
با پیشرفت سریع در دقت، کاهش هزینهها و افزایش مقیاسپذیری، RNA-Seq به استاندارد طلایی در مطالعات ترنسکریپتومیکس تبدیل شد.
دهه ۲۰۱۰: ترنسکریپتومیکس تکسلولی (scRNA-Seq)
در ادامهی این روند، دهه ۲۰۱۰ شاهد ظهور فناوری توالییابی RNA در سطح سلول منفرد یا scRNA-Seq بود.
این تکنولوژی به دانشمندان این امکان را داد تا:
-
بیان ژنها را در هر سلول بهصورت جداگانه اندازهگیری کنند.
-
ناهمگونی درونجمعیتی سلولها را کشف کنند، که در روشهای جمعی (bulk) مخفی میماند.
فناوریهایی مانند:
-
Smart-seq
-
Drop-seq
-
10x Genomics Chromium
امکان بررسی هزاران سلول منفرد در یک آزمایش واحد را فراهم کردند.
این پیشرفتها در حوزههایی مانند زیستشناسی رشد، ایمنیشناسی، علوم اعصاب و تحقیقات سرطان تحولی عظیم ایجاد کردند، چراکه درک تفاوتهای بین سلولی در این زمینهها بسیار حیاتی است.
فصل سوم: انواع RNA در ترنسکریپتوم
ترنسکریپتوم شامل مجموعهای متنوع از مولکولهای RNA است که از ژنوم رونویسی میشوند و هرکدام نقش خاصی در فرآیندهای سلولی ایفا میکنند. این RNAها بهطور کلی به دو دستهی اصلی تقسیم میشوند:
-
RNAهای کدکننده (Coding RNAs) که به پروتئین ترجمه میشوند.
-
RNAهای غیرکدکننده (Non-coding RNAs) که عملکرد دارند بدون اینکه ترجمه شوند.
درک انواع مختلف RNA برای شناخت جامعتر از ترنسکریپتومیکس ضروری است، چرا که هر نوع RNA، به شیوهای متفاوت در ساختار، عملکرد و تنظیم سلول نقش دارد.
RNA پیامرسان (mRNA)
RNA پیامرسان یا mRNA یکی از رایجترین و پرمطالعهترین انواع RNA در ترنسکریپتومیکس است.
این مولکول، میانجی مستقیم بین DNA و پروتئین محسوب میشود و اطلاعات ژنتیکی را از هسته به سیتوپلاسم منتقل میکند، جایی که فرآیند ترجمه (Translation) انجام میشود.
هر مولکول mRNA از یک ژن کدکنندهی پروتئین رونویسی میشود و شامل بخشهایی مانند:
-
کلاهک ۵′ (5’ Cap)
-
ناحیه کدکننده (Coding Region)
-
ناحیههای غیرترجمهای (UTRs)
-
دُم پلیآ (Poly-A Tail) در انتهای ۳′
این ساختارها در پایداری mRNA، محلیابی درون سلول، و کارایی ترجمه نقش حیاتی دارند.
میزان mRNA موجود در یک سلول، نمایانگر الگوی بیان ژنها در آن لحظه است؛ به همین دلیل، هدف اصلی در بسیاری از مطالعات ترنسکریپتومیکی محسوب میشود. فناوریهایی مانند RNA-Seq، میکروآری (Microarray) و qRT-PCR برای اندازهگیری سطح mRNA و بررسی تنظیم ژنها در شرایط مختلف زیستی، مانند رشد، استرس یا بیماریها به کار میروند.
با این حال، mRNA تنها کمتر از ۵٪ از کل RNA سلولی را تشکیل میدهد.
RNA ریبوزومی (rRNA)
RNA ریبوزومی یا rRNA، یکی از اجزای ساختاری و عملکردی اصلی ریبوزوم است، ماشینی که درون سلول مسئول سنتز پروتئینهاست.
در یوکاریوتها، ریبوزومها از چهار نوع rRNA (18S، 5.8S، 28S و 5S) همراه با دهها پروتئین ریبوزومی تشکیل میشوند.
این مولکولها، چهارچوب ساختاری ریبوزوم را شکل میدهند و همچنین فرآیند تشکیل پیوند پپتیدی را کاتالیز میکنند، بنابراین برای تولید پروتئین ضروریاند.
rRNA فراوانترین نوع RNA در سلول است و بیش از ۸۰٪ از کل RNA سلولی را تشکیل میدهد. به دلیل همین فراوانی، در آمادهسازی نمونههای RNA-Seq معمولاً rRNA بهصورت انتخابی حذف میشود تا امکان تحلیل RNAهای کمتر موجود مانند mRNA و ncRNA فراهم شود.
با اینکه rRNA یک مولکول خانهدار (Housekeeping RNA) محسوب میشود، تغییرات در بیان یا اصلاحات شیمیایی آن میتواند نشانهای از تغییرات متابولیسمی یا بیماریها باشد، بهویژه در سلولهایی که سریع تقسیم میشوند مانند سلولهای سرطانی.
RNA انتقالی (tRNA)
RNA انتقالی یا tRNA، نقشی کلیدی در ترجمه دارد. این مولکولها آمینو اسیدهای خاص را به زنجیرهی در حال رشد پلیپپتیدی در ریبوزوم منتقل میکنند.
هر tRNA دارای ساختار شبیه برگ شبدر (Cloverleaf) است و شامل:
-
حلقه آنتیکدون که با کدون متناظر در mRNA جفت میشود.
-
بازوی پذیرنده (Acceptor Arm) که آمینو اسید مناسب را به آن متصل میکند.
این ساختار، دقت در ساخت پروتئینها را تضمین میکند، بهطوری که هر بار یک آمینواسید بهدرستی وارد زنجیره میشود.
اگرچه tRNA نسبتاً کوچک است (حدود ۷۰ تا ۹۰ نوکلئوتید)، اما پس از رونویسی بهشدت اصلاح شیمیایی میشود و به ایزوفرمهای مختلفی وجود دارد.
علاوه بر نقش کلاسیک در ترجمه، قطعات حاصل از شکستن tRNA که به tsRNA (tRNA-derived small RNA) معروفند، در تنظیم ژنها، پاسخ به استرس و حتی فرایندهای سرطانزا نقش دارند.
در نتیجه، tRNAها بیش از آنچه قبلاً تصور میشد، در تنظیم عملکرد سلول فعال هستند.
RNAهای غیرکدکننده (ncRNA)
بخش در حال گسترش سریع در ترنسکریپتومیکس، مربوط به RNAهای غیرکدکننده یا ncRNA است—مولکولهایی که ترجمه نمیشوند ولی عملکردهای متنوع ساختاری و تنظیمی دارند.
این گروه به دو دستهی اصلی تقسیم میشود:
-
RNAهای کوتاه غیرکدکننده
-
RNAهای بلند غیرکدکننده (lncRNA)
میکروRNA (miRNA)
miRNAها مولکولهای RNA درونزاد و کوتاهی هستند (حدود ۲۲ نوکلئوتید) که بیان ژن را از طریق اتصال به mRNAهای هدف تنظیم میکنند. نتیجهی این اتصال معمولاً:
-
تخریب mRNA
-
یا مهار ترجمه است.
هر miRNA میتواند چندین ژن مختلف را هدف قرار دهد، و برعکس، هر ژن نیز میتواند تحت تنظیم چند miRNA باشد. این تعاملات، شبکههای تنظیمی پیچیدهای را تشکیل میدهند.
miRNAها در فرآیندهایی مانند رشد، آپوپتوز، پاسخ ایمنی و بهویژه در بیماریها مانند سرطان اهمیت بالایی دارند و بهعنوان نشانگرهای زیستی (Biomarkers) نیز بررسی میشوند.
RNAهای تداخلی کوچک (siRNA)
siRNAها از لحاظ اندازه و عملکرد شبیه miRNAها هستند، اما اغلب از منابع خارجی یا تجربی (Exogenous) منشأ میگیرند.
این RNAها، موجب هدایت کمپلکس خاموشسازی القایی RNA (RISC) به سمت mRNAهای مکمل شده و آنها را تجزیه میکنند.
siRNAها در پژوهشهای آزمایشگاهی و درمانی، برای خاموشسازی ژن (Gene Knockdown) و درمانهای مبتنی بر RNAi بسیار مورد استفاده قرار میگیرند.
RNAهای بلند غیرکدکننده (lncRNA)
lncRNAها، مولکولهای RNA طولانیتر از ۲۰۰ نوکلئوتید هستند که به پروتئین ترجمه نمیشوند.
این RNAها در عملکردهای گستردهای شرکت دارند:
-
بازآرایی کروماتین
-
تنظیم رونویسی
-
پردازش RNA
-
ایجاد چارچوب برای تجمع پروتئینها (Scaffolding)
lncRNAها اغلب دارای الگوهای بیان اختصاصی به بافت یا شرایط خاص هستند و به بیماریهایی مانند اختلالات عصبی و سرطان مرتبط هستند.
با توجه به پیچیدگی و تنوع بالا، lncRNAها هنوز حوزهای بسیار غنی برای اکتشافات بیشتر در ترنسکریپتومیکس هستند.
سایر انواع ncRNA
انواع دیگر RNAهای غیرکدکننده شامل:
-
piRNA (Piwi-interacting RNA): در حفظ پایداری ژنومی سلولهای زایشی نقش دارند.
-
snoRNA (Small Nucleolar RNA): راهنمای تعدیل شیمیایی rRNA و snRNAها هستند.
-
circRNA (RNA دایرهای): دستهای نسبتاً جدید که ممکن است نقش اسفنج miRNA یا تنظیمگرهای ژنی داشته باشند.
این RNAها لایههای پیچیدهتری از تنظیم در ترنسکریپتوم ایجاد میکنند.
فصل چهارم: تکنیکهای موجود در ترانسکریپتومیکس
مطالعهی ترانسکریپتومیکس به مجموعهای از تکنیکهای آزمایشگاهی و محاسباتی وابسته است که برای شناسایی، سنجش و تفسیر مولکولهای RNA در یک نمونه زیستی طراحی شدهاند. طی چند دههی اخیر، پیشرفتهای چشمگیر در زیستشناسی مولکولی و فناوری توالییابی، روشهای متعددی را در اختیار پژوهشگران قرار داده که هر یک دارای مزایا و محدودیتهای خاصی از نظر حساسیت، مقیاسپذیری، وضوح و هزینه هستند. انتخاب روش مناسب، وابسته به سؤال پژوهشی، نوع نمونه و منابع در دسترس است.
میکروآری (Microarrays)
میکروآریها از اولین ابزارهای پربازده (high-throughput) در تحلیل ترانسکریپتوم بودند. در این روش، RNA یا cDNA نشاندار شده با فلورسانس به سطحی جامد که هزاران پروب DNA تثبیتشده دارد، هیبرید میشود. شدت سیگنال فلورسانس در هر نقطه، میزان RNA هدف در نمونه را نشان میدهد. این فناوری در اواخر دهه ۱۹۹۰ و اوایل دهه ۲۰۰۰ انقلابی در مطالعات بیان ژن به وجود آورد، چرا که امکان سنجش همزمان هزاران نسخه RNA را فراهم کرد و به پژوهشگران اجازه داد الگوهای کلی بیان ژن را در شرایط مختلف بررسی کنند.
با وجود اهمیت تاریخی میکروآریها، این روش دارای محدودیتهایی است. مثلاً نیاز به دانش قبلی از توالی ژنها دارد، بنابراین قابلیت شناسایی نسخههای جدید یا تغییرات پیوندی (splicing) را ندارد. همچنین امکان برهمکنشهای غیراختصاصی (cross-hybridization) بین توالیهای مشابه وجود دارد که میتواند دقت دادهها را کاهش دهد. با این حال، میکروآریها همچنان گزینهای مقرونبهصرفه برای مطالعات مقایسهای در مقیاس بزرگ، به ویژه در موجودات مدل با ژنومهای شناختهشده، محسوب میشوند.
توالییابی RNA (RNA-Seq)
ظهور تکنولوژی توالییابی RNA یا RNA-Seq، ترانسکریپتومیکس را به سطحی کاملاً جدید برد. در این روش از فناوریهای توالییابی نسل جدید (NGS) برای تبدیل RNA به cDNA، قطعهقطعهسازی، و سپس توالییابی استفاده میشود. میلیونها خوانش کوتاه تولید شده با ژنوم مرجع همراستا میشوند یا در صورت نبود مرجع، به صورت de novo مونتاژ میگردند.
RNA-Seq دارای مزایای چشمگیری نسبت به میکروآری است:
-
میتواند نسخههای جدید، ایزوفرمهای حاصل از splice، فیوژنهای ژنی و RNAهای غیرکدکننده را شناسایی کند.
-
دادهها به صورت دیجیتال و کمی هستند و دامنه پویایی وسیعی دارند، یعنی میتوانند هم ژنهای بسیار بیانشده و هم ژنهای با بیان کم را شناسایی کنند.
این روش به استاندارد طلایی در مطالعات ترانسکریپتوم تبدیل شده، هرچند نیاز به پردازش دادههای حجیم، فضای ذخیرهسازی زیاد و هزینههای بالاتر دارد، بهویژه در پروژههایی با نمونههای زیاد.
واکنش زنجیرهای پلیمراز با زمان واقعی (qRT-PCR)
qRT-PCR یک روش بسیار حساس و دقیق برای سنجش بیان ژن است. در این روش ابتدا RNA به cDNA تبدیل شده، سپس با استفاده از پرایمرهای اختصاصی و رنگهای فلورسانس، فرآیند تکثیر در زمان واقعی دنبال میشود. شدت فلورسانس متناسب با مقدار اولیه RNA هدف در نمونه است.
هرچند qRT-PCR برای تحلیل جامع کل ترانسکریپتوم مناسب نیست، ولی روش استاندارد برای تأیید یافتههای RNA-Seq یا میکروآری محسوب میشود. این روش زمانی بسیار مفید است که تعداد کمی ژن مورد بررسی قرار دارند و دقت بالا مورد نیاز است. qRT-PCR سریع، مقرونبهصرفه و بهراحتی استانداردسازیپذیر است، و در تحقیقات پایه و تشخیصهای بالینی جایگاه مهمی دارد.
توالییابی RNA در سلول منفرد (scRNA-Seq)
روشهای سنتی ترانسکریپتومیکس، میانگین بیان ژنها در جمعیتی از سلولها را اندازهگیری میکردند، که این میتوانست ناهمگونی سلولی را پنهان کند. تکنولوژی توالییابی RNA در سلول منفرد (scRNA-Seq) این مشکل را حل کرده و امکان مطالعه بیان ژن در سطح تکسلول را فراهم آورده است. این موضوع در مطالعه بافتهای پیچیده که سلولهای مختلفی در کنار هم وجود دارند یا در مواردی که جمعیتهای نادری از سلولها نقش مهمی ایفا میکنند، حیاتی است.
scRNA-Seq شامل جداسازی سلولها (از طریق سیستمهای قطرهای مانند 10x Genomics یا روشهای پلیتمحور مانند Smart-seq) و سپس فرآیند لایز، تبدیل به cDNA، تقویت و توالییابی است. دادههای حاصل، امکان طبقهبندی سلولها بر اساس الگوهای بیان ژنی، ردیابی مسیرهای تمایزی و شناسایی پاسخهای سلولی دقیق به محرکها را فراهم میآورد.
با وجود چالشهایی چون مقدار کم RNA در هر سلول، نرخ افت بالا (dropout) و هزینهی بیشتر، scRNA-Seq به ابزاری کلیدی در حوزههایی مانند ایمونولوژی، نوروبیولوژی، زیستشناسی رشد و سرطانشناسی تبدیل شده است.
فصل پنجم: استخراج RNA و آمادهسازی کتابخانه (Library Preparation)
تحلیل دقیق و قابل اعتماد ترانسکریپتوم، با RNA با کیفیت بالا آغاز میشود. موفقیت تکنیکهای بعدی مانند RNA-Seq، میکروآری یا qRT-PCR تا حد زیادی به سلامت، خلوص و میزان RNA استخراجشده از نمونههای زیستی بستگی دارد. بنابراین، استخراج RNA و آمادهسازی کتابخانه توالییابی، مراحل ابتدایی بسیار حیاتی هستند که باید با دقت بهینهسازی و کنترل شوند. این فرآیندها نهتنها موفقیت فنی آزمایش را تعیین میکنند، بلکه تفسیر زیستی دادههای ترانسکریپتومیکس را نیز تحت تأثیر قرار میدهند.
استخراج RNA: اصول و روشها
استخراج RNA شامل جداسازی RNA از سلولها یا بافتهاست، بهگونهای که تخریب یا آلودگی آن با پروتئین، DNA یا مواد شیمیایی به حداقل برسد. RNA ذاتاً ناپایدار است، چرا که ساختاری تکرشتهای دارد و به راحتی توسط آنزیمهای ریبونوکلئاز (RNase) که در همه جا وجود دارند، تخریب میشود. بنابراین حفظ محیطی عاری از RNase در تمام مراحل استخراج، الزامی است. منابع رایج RNA شامل سلولهای کشتشده، بافتهای تازه یا فریز شده، خون و مایعات زیستی نظیر بزاق یا مایع مغزینخاعی هستند.
روشهای متنوعی برای استخراج RNA وجود دارد که هر یک با توجه به نوع نمونه، مزایای خاص خود را دارند. رایجترین روش، استخراج فنیل-کلروفرم است که معمولاً با معرفهای تجاری مانند TRIzol® انجام میشود. در این روش، RNA، DNA و پروتئینها بر اساس حلالیتشان به فازهای مجزا تفکیک میشوند. فاز آبی که RNA را در خود دارد، بهدقت جمعآوری شده و RNA با استفاده از الکلهایی مانند ایزوپروپانول یا اتانول، رسوب داده میشود. در مقابل، کیتهای مبتنی بر ستون سیلیکا یک روش سریعتر و راحتتر بهویژه در جریانهای کاری با بازده بالا هستند. این کیتها با ایجاد شرایط chaotropic، RNA را به سطح ستون متصل کرده و سپس از طریق مراحل شستوشو و الوت، RNA خالص را جدا میکنند.
صرفنظر از روش انتخابی، ارزیابی کیفیت RNA پیش از ادامه کار بسیار مهم است. سلامت RNA معمولاً با روشهای الکتروفورزی مانند Bioanalyzer یا TapeStation سنجیده میشود که عددی به نام RIN (عدد سلامت RNA) تولید میکنند. مقدار RIN بالای 7.0 برای بیشتر کاربردهای ترانسکریپتومیکس مناسب تلقی میشود. خلوص RNA با طیفسنجی (مانند NanoDrop) بررسی میشود؛ نسبتهای A260/A280 و A260/A230 نزدیک به ۲.۰ نشاندهنده آلودگی اندک هستند.
حذف DNA ژنومی و RNA ریبوزومی
یکی از چالشهای رایج هنگام استخراج RNA، آلودگی با DNA ژنومی (gDNA) است. حتی مقادیر اندک DNA میتواند دقت دادههای بیان ژن را—بهویژه در qRT-PCR و RNA-Seq—مختل کند. برای حل این مشکل، نمونهها با آنزیمهای DNase تیمار میشوند که DNA را تجزیه کرده و RNA را دستنخورده باقی میگذارند. بسیاری از کیتهای تجاری این مرحله را در پروتکل خود گنجاندهاند تا RNA عاری از DNA برای تحلیلهای بعدی فراهم شود.
در RNA-Seq، بهویژه زمانی که تمرکز بر روی mRNA (RNA پیامرسان) است، وجود مقدار فراوانی RNA ریبوزومی (rRNA) — که بیش از ۸۰٪ از کل RNA را تشکیل میدهد — میتواند مانع شناسایی نسخههای مورد نظر شود. برای رفع این مشکل، معمولاً از دو راهکار استفاده میشود: انتخاب poly(A) و حذف rRNA.
-
در روش انتخاب poly(A) از پرایمرهای oligo-dT یا مهرههای مغناطیسی برای جداسازی mRNAهای دارای دنباله پلیآدنین استفاده میشود که در نتیجه RNAهای کدکننده غنیسازی میشوند. البته این روش، RNAهای غیرپلیآدنیله (مانند بسیاری از RNAهای غیرکدکننده) را حذف میکند.
-
در مقابل، حذف rRNA با استفاده از پروبهای هیبریداسیون، rRNA را هدف قرار داده و حذف میکند. این روش امکان تحلیل طیف گستردهتری از RNAها را، از جمله RNAهای غیرکدکننده یا تخریبشده، فراهم میسازد.
انتخاب بین این دو روش به اهداف زیستی پژوهش و کیفیت RNA بستگی دارد.
آمادهسازی کتابخانه برای RNA-Seq
پس از بهدست آوردن RNA با کیفیت و غنیشده از نوع مناسب، گام بعدی آمادهسازی کتابخانه توالییابی (Library Preparation) است. هدف از این مرحله، تبدیل RNA به فرمتی سازگار با دستگاههای توالییابی است که شامل اتصال آداپتور و کدهای شاخص برای تکثیر و خوانش است. با وجود تفاوت در پلتفرمها، مراحل اصلی آمادهسازی کتابخانه عمدتاً مشابه هستند.
این فرآیند با شکستن RNA به قطعات کوچکتر (100 تا 300 نوکلئوتید) آغاز میشود. این کار از طریق هضم آنزیمی یا روشهای شیمیایی انجام میشود. در نمونههایی که از طریق انتخاب poly(A) غنی شدهاند، این قطعهقطعهسازی بعد از جداسازی mRNA انجام میشود. اما در روش حذف rRNA، ممکن است این مرحله مستقیماً بر روی RNA کامل انجام شود.
سپس RNA قطعهقطعهشده با استفاده از پرایمرهای تصادفی و آنزیم رونوشتبردار معکوس به cDNA (DNA مکمل) تبدیل میشود. این cDNA پایهای پایدار برای مراحل بعدی است.
در ادامه، رشته دوم DNA سنتز شده و cDNA دو رشتهای تولید میشود. سپس ترمیم انتها (End Repair)، اضافه کردن A در انتها (A-tailing) و اتصال آداپتورها (Adapter Ligation) انجام میگیرد. آداپتورها شامل توالیهای لازم برای اتصال به پلتفرم توالییابی و معمولاً بارکدهایی برای مولتیپلکس کردن نمونهها هستند. در پایان، کتابخانه با PCR تقویت میشود تا ماده کافی برای توالییابی فراهم شود. کیفیت و کمیت نهایی کتابخانه با روشهایی مانند Qubit (فلورومتری) و Bioanalyzer (تحلیل اندازه) بررسی میشود تا از یکنواختی و غلظت مناسب آن اطمینان حاصل گردد.
روشهای تخصصی در آمادهسازی کتابخانه
بسته به اهداف تحقیق، میتوان از استراتژیهای تخصصی در آمادهسازی کتابخانه استفاده کرد. به عنوان مثال، کتابخانههای جهتدار (Strand-Specific)، جهتگیری مولکولهای RNA را حفظ میکنند و امکان شناسایی دقیق RNAهای آنتیسنس و توضیح بهتر ژنهای همپوشان را فراهم میآورند.
پروتکلهای ترنسکریپت کامل مانند SMART-Seq، کل طول RNA را ثبت میکنند و بهویژه در مطالعات تکسلولی ارزشمند هستند. همچنین، میتوان پروتکلها را بهگونهای طراحی کرد که بر روی RNAهای خاص مانند miRNAها یا RNAهای حلقوی (Circular RNA) تمرکز کنند؛ این کار با مراحل انتخاب اندازه یا طراحی ویژه آداپتورها ممکن میشود.
فصل ۹: فناوریهایی که باعث پیشرفت ترنسکریپتومیکس شدهاند
رشته ترنسکریپتومیکس طی سالهای اخیر رشد چشمگیری داشته است که بخش عمدهای از آن به دلیل نوآوریهای فناورانهی قابلتوجهی است که توانایی ما را در اندازهگیری، تحلیل و تفسیر بیان RNA با دقت و وضوح بالا افزایش دادهاند. از روشهای اولیهی مبتنی بر هیبریداسیون تا فناوریهای پیشرفتهی توالییابی، این پیشرفتها نهتنها مقیاس و عمق مطالعات ترنسکریپتومی را گسترش دادهاند، بلکه باعث شدهاند این فناوریها در زمینههای مختلف پژوهشی و بالینی قابلدسترسیتر باشند. این فصل به بررسی مهمترین نقاط عطف فناورانهای میپردازد که مسیر ترنسکریپتومیکس را شکل دادهاند و نقش آنها را در تحول اکتشافات زیستی مورد بحث قرار میدهد.
میکروآری (Microarray): اولین گام مهم در ترنسکریپتومیکس
یکی از نخستین فناوریها در ترنسکریپتومیکس، میکروآری DNA بود. این پلتفرم به پژوهشگران اجازه داد تا سطوح بیان هزاران ژن را بهطور همزمان پایش کنند. میکروآریها بر پایهی اتصال RNA یا cDNA نشاندار شدهی فلورسنت به پروبهای DNA مکمل که روی سطحی ثابت نصب شدهاند عمل میکنند.
اگرچه این روش برای مطالعات ابتدایی بیان ژن بسیار تأثیرگذار بود، اما محدودیتهایی نیز داشت، از جمله:
-
نیاز به دانش قبلی از توالی ژنها
-
دامنهی دینامیکی نسبتاً پایین
با این حال، میکروآریها نقش بنیادینی در شکلگیری ترنسکریپتومیکس بهعنوان یک علم با توان بالا ایفا کردند و به دلیل هزینهی پایینتر و استانداردسازی بالا، هنوز در برخی کاربردهای تشخیصی و مقایسهای مورد استفاده قرار میگیرند.
RNA-Seq: جهشی انقلابی در تحلیل ترنسکریپتومی
ظهور فناوری توالییابی RNA یا RNA-Seq نقطهی عطفی در تحلیلهای ترنسکریپتومی محسوب میشود. برخلاف میکروآری، RNA-Seq به پروبهای از پیش طراحیشده نیازی ندارد، و این ویژگی امکان شناسایی بدون سوگیریِ ترنسکریپتهای شناختهشده و جدید را فراهم میکند.
این فناوری شامل تبدیل RNA به cDNA، قطعهقطعه کردن آن، و سپس توالییابی با استفاده از پلتفرمهای نسل جدید توالییابی (NGS) است.
مزایای RNA-Seq بسیار چشمگیرند، از جمله:
-
حساسیت بالا
-
دامنه دینامیکی گسترده
-
وضوح در سطح نوکلئوتید
-
توانایی شناسایی جایگزینیهای ژنی (alternative splicing)، ترنسکریپتهای ترکیبی (fusion transcripts)، و تغییرات پس از رونویسی
RNA-Seq اکنون به استاندارد طلایی در مطالعات ترنسکریپتومی تبدیل شده است و امکان درک عمیق و جامع از بیان ژن در شرایط، بافتها و گونههای مختلف را فراهم کرده است.
scRNA-Seq: انقلابی در ترنسکریپتومیکس تکسلولی
فناوری توالییابی RNA تکسلولی یا scRNA-Seq، زمینه را برای تحول بنیادی در مطالعهی بیان ژن در سطح تکسلول فراهم کرده است. این فناوری، مسئلهی ناهمگونی سلولی را هدف قرار میدهد که در بافتهای پیچیدهای مانند تومورها یا اندامهای در حال رشد اهمیت زیادی دارد.
scRNA-Seq شامل جداسازی سلولها، استخراج RNA آنها و توالییابی بهصورت مستقل از هر سلول است، که در نهایت منجر به ایجاد پروفایلی منحصر بهفرد از بیان ژن برای هر سلول میشود.
دستاوردهای این روش قابل توجهاند، از جمله:
-
شناسایی جمعیتهای نادر ولی مهم سلولی
-
ردیابی تبار سلولی در طول رشد و تکوین
-
نقشهبرداری از پاسخهای سلولی به محرکها یا بیماریها
ادغام scRNA-Seq با ترنسکریپتومیکس فضایی (spatial transcriptomics)، اطلاعات را غنیتر میکند، زیرا زمینهی فضایی بیان ژن در درون بافتها را حفظ میکند.
توالییابی با خوانش بلند (Long-Read Sequencing): دقت در ساختارهای پیچیدهتر
فناوریهای توالییابی با خوانش بلند که توسط پلتفرمهایی مانند Oxford Nanopore Technologies و Pacific Biosciences ارائه شدهاند، یک گام مهم دیگر محسوب میشوند. برخلاف پلتفرمهای خوانش کوتاه که نیازمند قطعهقطعه کردن RNA و بازسازی پیچیده آن هستند، این فناوریها قادرند مولکولهای RNA کامل را بهصورت مستقیم توالییابی کنند.
مزایای اصلی خوانش بلند شامل:
-
دقت بیشتر در شناسایی ایزوفرمها
-
کشف ساختارهای ترنسکریپتی پیچیده مانند ژنهای همپوشان و چند شکلی
اگرچه این روشها هنوز خروجی پایینتر و نرخ خطای بالاتری نسبت به خوانش کوتاه دارند، اما با بهبود مداوم فناوری، نقش آنها در مطالعات دقیقتر ترنسکریپتومی رو به افزایش است.
qRT-PCR: ابزار معتبر برای تأیید نتایج
روش qRT-PCR یا واکنش زنجیرهای پلیمراز با رونویسی معکوس کمی، همچنان بهعنوان ابزاری استاندارد برای تأیید یافتههای حاصل از روشهای با توان بالا مورد استفاده قرار میگیرد. این روش حساسیت و اختصاصیت بالایی دارد و برای تأیید بیان ژنهای خاص در نمونههای کوچک ایدهآل است.
همچنین در تشخیصهای بالینی کاربرد گستردهای دارد، مانند:
-
پایش بیماریهای باقیماندهی میکروسکوپی (MRD)
-
ارزیابی امضاهای ژنی مرتبط با پیشآگهی بیماری
ابزارهای محاسباتی و بیوانفورماتیک: کلید فهم دادههای پیچیده
فراتر از ابزارهای آزمایشگاهی، پیشرفت در ابزارهای محاسباتی و بیوانفورماتیکی نقش حیاتی در تجزیه و تحلیل دادههای عظیم ترنسکریپتومی ایفا کرده است.
امکانات این حوزه شامل:
-
الگوریتمها و مدلهای آماری پیشرفته
-
روشهای یادگیری ماشین برای تطبیق، کمّیسازی و تحلیل الگوهای بیان ژن
-
شبکهسازی تنظیمی و پیشبینی عملکرد ژنها
علاوه بر آن، پردازش ابری و پایگاههای دادهی دسترسی آزاد به پژوهشگران کمک میکند تا به مجموعه دادههای بزرگ دست یابند، همکاریهای پژوهشی گسترده ایجاد کنند و تحلیلهای ترکیبی (meta-analysis) انجام دهند که پیشتر ممکن نبود.
یکپارچهسازی مولتیاُمیک: گامی به سوی زیستشناسی سامانهای
ظهور یکپارچهسازی دادههای مولتیاُمیک (multi-omics) که ترکیب اطلاعات ترنسکریپتومی با دادههای ژنومی، پروتئومی، اپیژنومی و متابولومی است، مرزهای علم زیستشناسی را جابجا کرده است.
فناوریهایی که این یکپارچهسازی را ممکن کردهاند—مانند پلتفرمهای چند-اُمیکی تکسلولی—امکان ساخت مدلهای جامع از عملکرد و تنظیمات سلولی را فراهم میکنند. این نگاه جامع برای درک بیماریهای پیچیده و پیشبرد پزشکی دقیق (precision medicine) ضروری است.
جمعبندی: آیندهای روشن برای ترنسکریپتومیکس
تحول فناوری در ترنسکریپتومیکس مرزهای زیستشناسی مولکولی را دگرگون کرده است. از تحلیلهای حجمی (bulk) تا دقت در سطح تکسلول، و از خوانشهای کوتاه تا توالییابی کامل مولکولی، هر پیشرفت، لایهای جدید از عمق، دقت و اهمیت زیستی را به این علم افزوده است.
با بلوغ بیشتر این فناوریها و همگرایی آنها، میتوان انتظار داشت که درک ما از تنظیم ژنی، تنوع سلولی و مکانیسمهای بیماری به شکل چشمگیری ارتقاء یابد—و ترنسکریپتومیکس به ستون اصلی علوم زیستی مدرن تبدیل شود.
فصل ۱۱: ادغام ترنسکریپتومیکس با دیگر رشتههای اومیکس
با پیشرفت روزافزون درک علمی ما از سیستمهای زیستی که به صورت چندبعدی مطرح میشوند، ادغام ترنسکریپتومیکس با دیگر رشتههای “اومیکس” به عنوان یک گام مهم برای رسیدن به دیدگاهی جامع از زندگی در سطح مولکولی مطرح شده است. در حالی که ترنسکریپتومیکس الگوهای بیان RNA را نشان میدهد که منعکسکننده استفاده فعال ژنها هستند، اما تنها یک لایه از تنظیمات سلولی به شمار میرود. ترکیب دادههای ترنسکریپتومیک با اطلاعات مکملی از ژنومیکس، پروتئومیکس، متابولومیکس و اپیژنومیکس به محققان این امکان را میدهد تا شبکههای پیچیده تنظیمی را کشف کنند، روابط علّی را دنبال کنند و مدلهای پیشبینیکنندهتری از سلامت و بیماری توسعه دهند. این فصل بررسی میکند که چگونه ترنسکریپتومیکس در چشمانداز گستردهتر چند-اومیکس جای میگیرد و مزایا، استراتژیها و چالشهای این رویکرد بینرشتهای را برجسته میکند.
ژنومیکس و ترنسکریپتومیکس به طور طبیعی به هم مرتبط هستند. ژنومیکس نقشهی ثابت یک موجود زنده است — کل توالی DNA و تمامی عناصر کدکننده و تنظیمی آن — در حالی که ترنسکریپتومیکس نشان میدهد که کدام بخشهای این نقشه در هر لحظه به RNA تبدیل میشوند. ادغام این دو داده به دانشمندان اجازه میدهد که واریانتهای ژنتیکی مانند پلیمورفیسمهای تک نوکلئوتیدی (SNP) یا تغییرات ساختاری را با تغییرات در بیان ژن مرتبط کنند. تحلیل eQTL (لکوسهای صفات کمی بیان) نمونه برجستهای از این ادغام است که به پژوهشگران کمک میکند مناطقی از ژنوم را شناسایی کنند که سطح بیان RNA را تحت تأثیر قرار میدهند. این اطلاعات برای درک اینکه چگونه تفاوتهای ژنتیکی ارثی به حساسیت به بیماری و تنوع فنوتیپی کمک میکنند، حیاتی است.
پروتئومیکس که بر شناسایی و کمیسازی پروتئینها تمرکز دارد، بعد حیاتی دیگری به دادههای ترنسکریپتومیک اضافه میکند. چون پروتئینها مولکولهای عملکردی سلول هستند، ارتباط سطح RNA با مقدار پروتئین تصویر کاملتری از فعالیت ژن ارائه میدهد. اما سطح mRNA همیشه نمیتواند سطح پروتئین را پیشبینی کند، زیرا تفاوتهایی در کارایی ترجمه، پایداری پروتئین و تغییرات پس از ترجمه وجود دارد. مطالعات ترکیبی که RNA-Seq را با پروتئومیکس مبتنی بر طیفسنجی جرمی جفت میکنند، به پر کردن این فاصله کمک میکنند و نشان میدهند کدام ترنسکریپتها ترجمه میشوند و چگونه بیان پروتئین در شرایط خاص تغییر میکند. این رویکرد به درک گلوگاههای تنظیمی کمک میکند و لایههای کنترل فراتر از رونویسی را آشکار میسازد.
متابولومیکس، مطالعه متابولیتهای کوچک مولکولی، بینشی درباره نتایج بیوشیمیایی بیان ژن فراهم میکند. در حالی که ترنسکریپتومیکس میگوید کدام ژنها فعال هستند، متابولومیکس نشان میدهد این بیان چگونه در متابولیسم سلولی تأثیر میگذارد. ادغام این دو نوع داده به پژوهشگران امکان میدهد برنامههای رونویسی را شناسایی کنند که تغییرات در مسیرهای متابولیکی را هدایت میکنند. این ادغام به ویژه در حوزههایی مانند زیستشناسی سرطان که تغییرات در بیان ژن اغلب منجر به تغییر در متابولیسم انرژی میشود، و در تغذیه و داروشناسی که بیان ژن بر متابولیسم دارو یا استفاده از مواد مغذی تأثیر دارد، بسیار مفید است.
اپیژنومیکس تغییرات شیمیایی در DNA و هیستونها را بررسی میکند که بیان ژن را بدون تغییر در توالی DNA تنظیم میکنند. این تغییرات شامل متیلاسیون DNA، استیلاسیون هیستون و بازسازی کروماتین هستند که میتوانند دسترسی به رونویسی را تنظیم کنند. ادغام دادههای ترنسکریپتومیک و اپیژنومیک به دانشمندان کمک میکند تا بفهمند وضعیتهای اپیژنتیکی چگونه فعالیت ژن را تحت تأثیر قرار میدهند. به عنوان مثال، با مقایسه مناطق کروماتین باز (با استفاده از آزمایشهایی مانند ATAC-seq) با پروفایلهای بیان ژن، میتوان عناصر تنظیمی مانند افزایندهها و پروموترهای فعال در شرایط خاص را شناسایی کرد. این رویکرد ادغامی در زیستشناسی توسعه و مطالعه تأثیرات محیطی بر تنظیم ژن اهمیت زیادی دارد.
ظهور فناوریهای چند-اومیکس تکسلولی پیشرفت قابل توجهی در این زمینه است که امکان شناسایی همزمان چندین لایه مولکولی از یک سلول منفرد را فراهم میکند. این پلتفرمها میتوانند ترنسکریپتومها را همراه با دسترسی کروماتین (مثلاً scRNA-seq + scATAC-seq)، متیلاسیون DNA یا پروتئینهای سطحی (مثلاً CITE-seq) اندازهگیری کنند. این پروفایل جامع به پژوهشگران اجازه میدهد تا ناهمگنی سلولی را با دقت بینظیری بررسی کنند و مدلهای دقیقی از تمایز خط سلولی، پاسخهای ایمنی و پیشرفت بیماری بسازند. قابلیت پیوند چندین نوع داده از همان سلول، اثرات مخدوشکننده میانگینگیری جمعیت سلولی را حذف کرده و مسیرهای جدیدی برای پزشکی شخصی باز میکند.
با وجود قدرت ادغام چند-اومیکس، این رویکرد بدون چالش نیست. تفاوت در نوع دادهها، مقیاسها و میزان نویز، تراز و تفسیر دادهها را پیچیده میکند. هر لایه اومیکس نیازمند آمادهسازی، توالییابی و روشهای تحلیلی خاص خود است و ادغام آنها اغلب نیازمند چارچوبهای آماری و محاسباتی پیشرفته است. تلاشهایی برای توسعه روشهای استاندارد و نرمافزارهای قابل تعامل برای مدیریت پیچیدگی دادههای چند-اومیکس در جریان است. همچنین، یادگیری ماشینی و هوش مصنوعی به طور فزایندهای برای شناسایی الگوها، استنباط شبکههای علّی و پیشبینی نتایج فنوتیپی از دادههای ادغام شده استفاده میشوند.
در پایان، ادغام ترنسکریپتومیکس با دیگر رشتههای اومیکس به یک استراتژی حیاتی برای دستیابی به درک سیستممحور از زیستشناسی تبدیل شده است. این دیدگاه چندلایه به پژوهشگران امکان میدهد جریان اطلاعات از ژنوتیپ به فنوتیپ را دنبال کنند و شبکههای پیچیدهای که زیربنای عملکرد و اختلال سلولی هستند را کشف کنند. با پیشرفت فناوریها و ابزارهای تحلیلی، ادغام چند-اومیکس نوید میدهد که درک ما را از توسعه، مکانیزمهای بیماری و پاسخهای درمانی عمیقتر کند و راه را برای رویکردهای جامعتر و فردیسازی شده در زیستشناسی و پزشکی هموار سازد.